图像处理笔记(一)
直方图均衡化
参考文档
[1] Jan Erik Solem. Python计算机视觉编程 (图灵程序设计丛书) (p. 11). 人民邮电出版社.
[2] 直方图均衡化
代码
1 | # 直方图均衡化 |
讲解
解决的问题
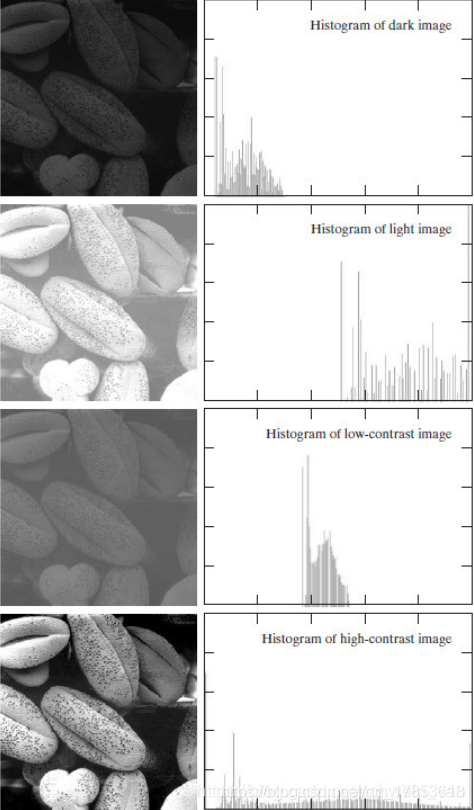
通常,暗图像直方图的分量集中在灰度较低的一端,而亮图像直方图分量偏向于灰度较高的一端。
从图中可以得到这样的结论:如果一幅图像的灰度直方图几乎覆盖了整个灰度的取值范围,并且除了个别灰度值的个数较为突出,整个灰度值分布近似于均匀分布,那么这幅图像就具有较大的灰度动态范围和较高的对比度,同时图像的细节更为丰富。已经证明,仅仅依靠输入图像的直方图信息,就可以得到一个变换函数,利用该变换函数可以将输入图像达到上述效果,该过程就是直方图均衡化。
一句话说,就是直方图的值都聚在一块不清楚,均衡化之后能清除很多。
方法
将图像中的灰度级均匀的映射到整个灰度级范围,具体的方法如下:
- 前提假设:在一张有N个像素点的图片中,有灰度值的取值范围是0~M,你希望映射到的范围是\([x,y]\)
- 第一步:统计每一个灰度值出现的次数\(t_i\),其中\(i\)代表灰度,\(t_i\)代表其出现的次数
- 第二步:计算每一种灰度值对应的像素点个数在总体中占比\(P_i = \frac{t_i}{N}\)
- 第三步:计算累计占比\(\sigma P_i=\Sigma_{i=0}^{i}P_i\)
- 第四步:计算映射后的灰度值\(final_i=\sigma P_i*(y-x+1)\)
图像平均
代码
1 | def compute_average(imList): |
讲解
没啥好讲的,把一堆图拉到一块,平均一下即可。这个方法可以用于降噪, 但是我们也会发现,这个方法要把所有图片都拉到内存里,如果要平均很多的图片的话,我们需要拉很多的内存,不过又有一种方法就是可以每次平均一张也就是每次新引入一张,也可以。
图像的主成分分析(PCA)
参考资料
[1] Jan Erik Solem. Python计算机视觉编程 (图灵程序设计丛书) (p. 11). 人民邮电出版社.
[2] 机器学习经典算法:PCA降维与SVD矩阵分解—— 自兴人工智能教育
主成分分析法,老朋友了。这个目前已知的有两种用法:
- 降低数据维度
- 求每一个值在总体中的客观权重(重要程度)
利用这个可以做图像的缩略图,它的优点是能在降维的时候,同时尽量多的保持训练数据的信息,这就很nb了。
代码
1 | # PCA |
原理
PCA
PCA做了什么?
众所周知,PCA可以用来降维,举例来说,如果现在由一组数据,规格是100*4的,也就是说,有100组数据,每组数据有4个特征,现在我们想把它的每一组特征的数目降到2,变化完成后,变成100*2的矩阵,那么这个变化需要什么呢?学过线代的同学都懂了,这里需要在乘上个4*2的矩阵就可以完成这种变化,那么求这个4*2的矩阵,就是PCA的主要任务。
操作流程
第一步:先对数据进行标准化操作
第二步:计算协方差矩阵
什么是协方差:
协方差:\(\sigma_{jk}=\frac{1}{n-1}\Sigma_{i=1}^n(x_{ij}-x_{j}.mean)(x_{ik}-x_k.mean)\)
协方差代表着两个数据之间的相关性,相关性越大,协方差越大
协方差矩阵蕴含着任意两个变量之间的协方差
协方差矩阵的计算:\(\Sigma=\frac{1}{n-1}((X-x.mean^T(X-x.mean))\)
对协方差矩阵求特征值和特征向量:
什么是特征值和特征向量?
线性代数学了一年多了,现在只记得加减乘和转置了,取个逆都是高端操作了,特征值和特征向量是啥来着?不记得了。
直接看定义:\(A\) 是\(n\)阶方阵,若存在数\(\lambda\)和非零向量x使得\(Ax = \lambda x\)那么称\(\lambda\)是A的一个特征值,\(x\)为A的对应于特征值\(\lambda\)的特征向量。
我们现在有一个这样的矩阵: \[ a = \left[ \begin{matrix} 1 & 2&3&4\\ 2&1&5&6\\ 3&5&1&7\\ 4&6&7&1 \end{matrix} \right] \] 我们使用语句:np.linalg.eig(a),得到了以下结果
1
2
3
4
5
6
7
8
9In [9]: a = np.array([[1,2,3,4],[2,1,5,6],[3,5,1,7],[4,6,7,1]])
In [10]: b,c = np.linalg.eig(a)
In [11]: b, c
Out[11]:
(array([15.01698746, -0.67469831, -4.04123361, -6.30105553]),
array([[ 0.35249192, 0.89949656, -0.21038586, -0.14964351],
[ 0.49287878, -0.40507255, -0.72842863, -0.24975682],
[ 0.54203206, -0.15251679, 0.63351659, -0.53065677],
[ 0.58225527, -0.05967247, 0.15422794, 0.79600989]]))我们根据刚才的定义进行检验:
1
2
3
4In [26]: b[0]*c[:,0]
Out[26]: array([5.29336674, 7.40155453, 8.13968862, 8.74372006])
In [27]: np.dot(a,c[:,0])
Out[27]: array([5.29336674, 7.40155453, 8.13968862, 8.74372006])1
2
3
4
5In [28]: b[1]*c[:,1]
Out[28]: array([-0.60688881, 0.27330177, 0.10290282, 0.04026092])
In [29]: np.dot(a,c[:,1])
Out[29]: array([-0.60688881, 0.27330177, 0.10290282, 0.04026092])所以我们得到了四组特征向量和特征值,据说特征值代表着当前特征向量的重要程度。
对这些特征值进行归一化,将他们映射到1~100的区间中。
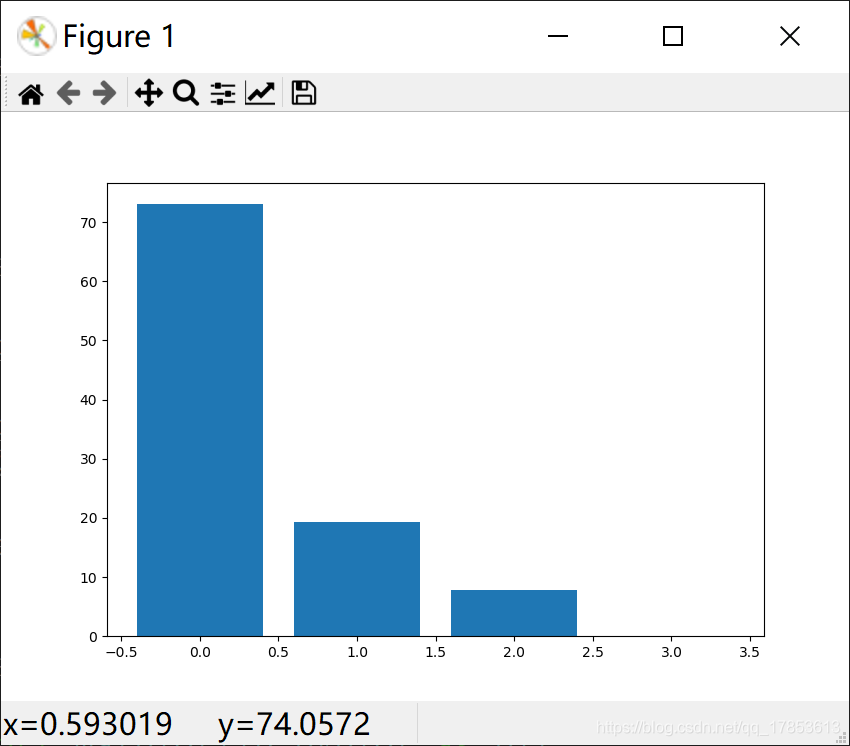
可以看到,后两个特征值都太小了,而且我们的任务是把这个100*4的矩阵转化成100*2的矩阵,所以我们需要选取两个特征向量,所以呢我们就去选最大的两个特征值所对应的特征向量,也就是特征向量0和1。
将他们合成一个4*2的矩阵,计算原矩阵与他们的矩阵乘法,就可以得到新的特征矩阵了。
SVD矩阵分裂
目标
将一个矩阵A分解三个矩阵的乘积:
如下:
graph LR A[A m*n] -->u[u m*m] A[A m*n] -->S[S m*n] A[A m*n] -->V[V n*n]
我们的目标是将一个m*n的矩阵A拆分成u*S*V的形式,他们的形状如上图所示。
其中S是一个对角矩阵,对角矩阵就是只有主对角线上的元素不是零的矩阵,你还看不懂什么叫对角矩阵的话建议百度。
剩下的看这篇博客 https://mp.weixin.qq.com/s/Dv51K8JETakIKe5dPBAPVg
使用pickle模块
pickle可以接受几乎所有python的对象,并且将其转换成字符串表示,这个过程叫做封装(pickling)。从字符串表示中重构该对象,成为拆封(unpickling)。这些字符串表示可以方便的储存和传输。
储存
1 | f = open('font_pca_modules.pkl', 'wb') |
在上述的例子中,许多对象可以保存到同一文件中。pickle模块中有很多不同协议可以生成.pkl文件,如果不确定的话,最好以二进制文件的形式去读取、写入。在其他python会话中载入数据,只需要使用load()方法。
读取
1 | f = open('font_pca_modules.pkl', 'rb') |
需要注意的是,dump的顺序和load的顺序必须一样,当然,与文件相关的读写操作可以用with语句完成,这样可以避免文件开关导致的错误。
使用with的保存和载入
保存
1 | with open('font_pca_modules.pkl', 'wb') as f: |
载入
1 | with open('font_pca_modules.pkl', 'rb') as f: |
从本质上说这两种方法没什么不同的,就是在with的作用域中,f是开的,出了with的作用域后,f就会自动关闭,当然我本人是不喜欢这种代码形式的,但是为了让自己的代码风格尽量规范,我决定适应这一形式。
Scipy
图像模糊
作者在这里又不经意的抛出了一个重点。。
图像的高斯模糊的实质就是灰度图像I和一个高斯核进行卷积的操作。
这里给没有入门的兄弟萌讲一下,什么叫卷积,数学上两个函数的卷积,我目前还没有学(大二下)。图的卷积只是借了一个概念而已,简单地讲:假如有一个\(n \times n\)的矩阵A ,还有一个\(3 \times3\)的滤波器B。不要纠结什么叫滤波器,下面会讲清楚的。他们现在分别长这个样子: \[ A = \left[ \begin{matrix} a_{1,1} & a_{1,2} &... & a_{1,n} \\ a_{2,1} & a_{2,2} &... & a_{2,n} \\ ... &...& &... \\ a_{n,1} &a_{n,2}&... &a_{n,n} \end{matrix} \right], B = \left[ \begin{matrix} -1&0&1\\ -1&0&1\\ -1&0&1 \end{matrix} \right] \] A和B进行卷积操作,生成了个什么玩意呢?很简单,生成了一个新的矩阵,这个矩阵的每一个元素就像是下面的图片里一样(特殊说明:下面的图片是我在这个网站上面直接拿的)
所以我们可以做一个联想,卷积就是拿着一个放大镜把这个图扫一遍生成一张新的图,放大镜(也就是滤波器)不一样,得到的结果也不一样。
下面我们来看一下如何对一张图片进行高斯模糊。talk is free,代码来了:
1 | from PIL import Image |
主体就只有下面两行,filters.gaussian_filter(im, 5),这里的第一个参数代表需要进行高斯模糊的矩阵,第二个代表标准差,这个大家都懂,最后的效果如下:

我们将标准差从5降低到2进行观察,发现果然模糊的程度降低了。

如果要对一张彩色图像进行高斯模糊(彩色图形就是多通道图形,我们的灰度图只有一个通道,而通常彩色图形是由RGB三个通道组成的),我们只需要对他的每一层进行高斯模糊即可。
1 | from PIL import Image |
效果如下:
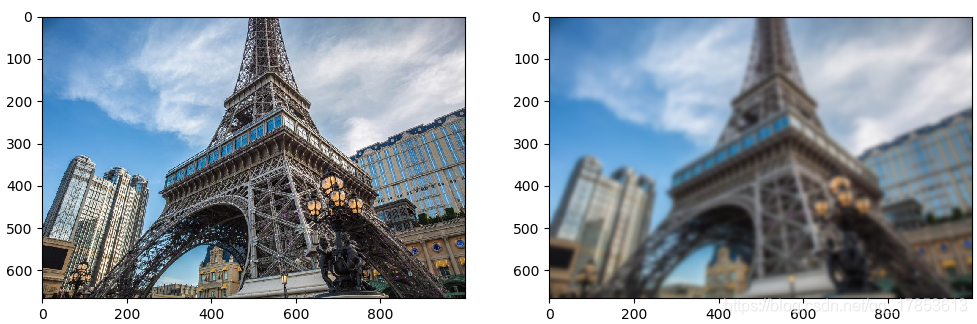
将标准差降低到2,效果如下

对于三通道,我产生了一种很奇葩的想法, 那就是如果每一层用于模糊的标准差不一样会发生什么?我将模糊的值从全都是五改为了i*3:
1 | im2[:, :, i] = filters.gaussian_filter(im[:, :, i], i*3) |
模糊后的图片变成了这样:

我们可以理解,因为图像中的红色被模糊的较少,比较强烈,而蓝色和绿色被模糊的程度较大,无法保持原来的亚子,所以红色的框架被显现出来。
将模糊值调节到i*20,我们会发现, 这变成了一张具有魔幻现实主义的图片😂

图像导数
参考资料
[1] 边缘检测的各种微分算子比较
从书中可以看出来,在很多应用中图像强度的变化情况是非常重要的信息。强度的变化可以用灰度图像\(I\)的\(x\)和\(y\)方向导数\(I_x\)和\(I_y\)进行描述。
梯度之中包含两个信息:
梯度的大小:
\(|\Delta I| = \sqrt{I_x^2 + I_y^2}\)
他描述了图像变化的强弱
梯度的角度:
\(\alpha = arctan2(I_y,I_x)\)
描述了图像中在每一个点上强度变化的最大方向。
我们可以使用离散点近似的方式来计算图像的导数。图像的导数可以通过卷积简单地实现: \[ I_x = I * D_x ,I_y = I*D_y \] 对于\(D_x\)和\(D_y\),通常选择 Prewitt 滤波器: \[ D_x = \left[ \begin{matrix} -1&0&1\\ -1&0&1\\ -1&0&1 \end{matrix} \right], D_y = \left[ \begin{matrix} -1&-1&-1\\ 0&0&0\\ 1&1&1 \end{matrix} \right] \] 或者是 Sobel 滤波器: \[ D_x = \left[ \begin{matrix} -1&0&1\\ -2&0&2\\ -1&0&1 \end{matrix} \right], D_y = \left[ \begin{matrix} -1&-2&-1\\ 0&0&0\\ 1&2&1 \end{matrix} \right] \]
如果你看懂了上一节像关于什么是滤波器的问题的话,这里应该不难理解,如果你没看懂,那大概率是我没有讲清楚,你可以去网上搜集一些视频教程或是博客帮助自己理解。这两种滤波器的区别是: 据经验得知Sobel要比Prewitt更能准确检测图像边缘。
下面看一下效果:
1 | from PIL import Image |

上面展示的是最后提取的X轴和Y轴综合的梯度图
 上面两张分别是X轴和Y轴检测的。
上面两张分别是X轴和Y轴检测的。
今天先到这里。